IPC分类号 : C12N9/88,C12N15/60,C12N15/70,C12N1/21,C12P5/00,C12R1/19
专利摘要
本发明公开了一种油楠倍半萜合成酶及其编码基因和应用。本发明提供了一种新的倍半萜合成酶——油楠倍半萜合成酶SgSTPS1及其编码基因,油楠倍半萜合成酶SgSTPS1蛋白在原核表达后可生成活性的倍半萜合成酶,催化底物FPP产生β‑石竹烯、异石竹烯和α‑石竹烯,可用于其大量生产。
权利要求
1.油楠倍半萜合成酶SgSTPS1在制备β-石竹烯、异石竹烯和/或α-石竹烯中的应用,所述的油楠倍半萜合成酶SgSTPS1,其氨基酸序列如SEQ ID NO.2所示。
2.根据权利要求1所述的应用,其特征在于,是以法呢基焦磷酸为底物,经油楠倍半萜合成酶SgSTPS1催化产生β-石竹烯、异石竹烯和α-石竹烯。
说明书
技术领域
本发明属于植物基因工程技术领域,具体涉及一种油楠倍半萜合成酶及其编码基因和应用。
背景技术
油楠(Sindora glabra Merr.ex de Wit)隶属于苏木科油楠属,原生分布于我国海南省,为热带和南亚热带高大乔木,是国家二级重点保护野生植物。油楠最重要的特征是其树干木质部受损后可分泌出树脂油,其可燃性与柴油相似,故又称“柴油树”,在油用、药用、观赏和材用等方面具有较广阔的开发利用前景。
油楠树脂油成分主要包括倍半萜类,约占70%以上,以及二萜类,约14%以上,其中α-可巴烯、β-石竹烯和δ-杜松烯三者含量占倍半萜类的2/3以上。油楠油亦含活性成分α-紫穗槐烯、荜澄茄油烯、α-蛇麻烯、环苜蓿烯等,进一步,油楠油亦含有挥发性成分大牻牛儿烯、α-香橙烯、δ-杜松醇、α-檀香醇和α-依兰油烯等代表香气的主要成分,在开发精油方面展现了巨大的开发潜力。倍半萜类化合物是天然药物化学中研究较为活跃的领域之一,具有抗肿瘤、抑菌等活性。然而,关于油楠中萜类合成酶的生物合成基因还未被分离和鉴定。β-石竹烯被广泛应用于食品香料香精等产品中,但其生产主要是通过直接从植物中提取进行,而未见采用生物工程方法生产的报道。
发明内容
本发明的目的是针对现有技术的不足,提供一种新的倍半萜合成酶及其编码基因以及倍半萜合成酶的应用。
本发明的第一个目的是提供一种油楠倍半萜合成酶SgSTPS1,其氨基酸序列如SEQID NO.2所示。
本发明的第二个目的是提供所述的油楠倍半萜合成酶SgSTPS1的编码基因。
优选,所述的编码基因,其核苷酸序列如SEQ ID NO.3所示。
优选,所述的编码基因,其核苷酸序列如SEQ ID NO.1所示。
与所述的油楠倍半萜合成酶SgSTPS1氨基酸具有50%以上相似性的序列,或与其具有相同功能的保守性变异多肽、活性片段或活性衍生物,例如,将氨基酸序列经过一个或多个取代、缺失或添加而形成的氨基酸序列也属于本发明的保护范围。与所述的油楠倍半萜合成酶SgSTPS1的编码基因的核苷酸序列具有50%以上相似性的序列也属于本发明的保护范围。
本发明还提供一种含有所述的油楠倍半萜合成酶SgSTPS1的编码基因的重组表达载体。
所述的表达载体,优选为表达载体pET-30a。
本发明还提供一种含有所述的油楠倍半萜合成酶SgSTPS1的编码基因的基因工程菌。
所述的基因工程菌,优选为大肠杆菌Top10或大肠杆菌BL21(DE3)。
本发明的第三个目的是提供所述的油楠倍半萜合成酶SgSTPS1在制备β-石竹烯、异石竹烯和/或α-石竹烯中的应用。
优选,所述的应用,是以法呢基焦磷酸(FPP)为底物,经油楠倍半萜合成酶SgSTPS1催化产生β-石竹烯、异石竹烯和α-石竹烯。
本发明提供了一种新的倍半萜合成酶——油楠倍半萜合成酶SgSTPS1及其编码基因,该蛋白在原核表达后可生成活性的倍半萜合成酶,催化底物FPP产生β-石竹烯、异石竹烯和α-石竹烯,可用于其大量生产。
附图说明
图1是目的基因PCR片段。
图2是目的基因菌落PCR电泳图。
图3是目的蛋白SgSTPS1的SDS-PAGE检测。
图4是SDS-PAGE分析目的蛋白SgSTPS1上清纯化结果。
图5是目的蛋白SgSTPS1的SDS-PAGE和Western-blot检测。
图6是GC-MS检测图谱。
具体实施方式
以下实施例是对本发明的进一步说明,而不是对本发明的限制。以下实例中未具体注明的实验方法,均可按照常规方法进行,或按照所用产品生产厂商的使用说明;所使用的材料、试剂等,如无特殊说明,均可通过商业途径得到。
实施例1
1.克隆油楠倍半萜合成酶基因SgSTPS1,构建克隆载体及转化原核细胞
提取油楠茎部组织的RNA,采用逆转录酶M-MLV,逆转录反应合成cDNA。以该cDNA为模板,正向引物为:5'-ATGGGGCGACCCACGGCAA-3',反向引物为:5'-CTATTTGTTGACATCAATGGC-3',利用TakaRa公司Ex Taq DNA聚合酶,进行PCR扩增;PCR条件为:94℃5min;94℃30s,55℃1min,72℃1min30s,35个循环;72℃延伸10min。PCR产物用1%的琼脂糖凝胶电泳检测,结果如图1所示,图1的M为DNA marker,箭头为目的基因SgSTPS1片段。
采用琼脂糖凝胶电泳胶回收试剂盒的方法回收目的基因片段,将目的片段进行TA克隆,连接到载体Pmd-18TA克隆载体中,然后将其转化到大肠杆菌Top10克隆菌株中,转化条件为:在200μL感受态细胞中加入5μL连接产物,轻轻混匀后冰浴30min;迅速放入42℃水浴中热激90s,立即置于冰上2min;加入800μL LB培养基,37℃慢摇培养1h;将菌液6000rpm离心2min,弃600μL上清,悬浮菌体,涂布于含有抗生素(Amp)的LB平板上,37℃倒置暗培养12~16h。采用菌落PCR进行阳性克隆筛选,筛选方法为:从转化平板上随机挑取单菌落,置于1.5mL离心管中的液体培养基中培养。将每管进行编号,各管取1μL用作模板进行PCR检测,剩余培养物保存于4℃,检测为阳性的菌落于平板或甘油管保存备用。PCR反应体系为:1μL菌液,正向引物5'-CGCCAGGGTTTTCCCAGTCACGAC-3',反向引物5'-AGCGGATAACAATTTCACACAGGA-3',1x Buffer,0.25mM dNTP mixture,0.5μL ExTaq DNA聚合酶,混合后进行PCR扩展。PCR程序为:94℃5min;94℃30s,55℃1min,72℃1min30s,35个循环;72℃延伸10min。菌落PCR结果如图2所示,图2的M为DNAmarker,1、2和7为阳性菌。挑选阳性单克隆菌落,提取质粒后送交测序。经测序分析,克隆得到的倍半萜合成酶基因SgSTPS1,其核苷酸序列如SEQ ID NO.1所示,其含有1650个碱基,编码的蛋白命名为倍半萜合成酶SgSTPS1,共549个氨基酸,具体氨基酸序列如SEQID NO.2所示。并由此得到将倍半萜合成酶基因SgSTPS1插入到Pmd-18TA克隆载体的重组质粒成功转化至大肠杆菌Top10的阳性菌,命名为Top10-SgSTPS1。
2、油楠倍半萜合成酶基因SgSTPS1密码子优化和全基因合成、构建表达载体及转化原核细胞
采用密码子优化软件对表达SgSTPS1蛋白的密码子进行优化,优化后的编码基因的碱基序列如SEQ ID NO.3所示。采用全基因合成方法合成密码子优化后的倍半萜合成酶基因SgSTPS1全长序列。通过限制性酶切位点Nde I和Hind III对优化的SgSTPS1基因和质粒pET30a分别进行双酶切,酶切体系为:Nde I和Hind III各1μL,基因浓度为0.3μg,质粒浓度为1μg,加入灭菌的双蒸水至30μL,酶切时间为1h。酶切后利用核酸纯化回收试剂盒纯化回收得到经过双酶切的优化的SgSTPS1基因片段和pET30a载体。将酶切后的优化的SgSTPS1基因片段连接至酶切后的表达载体pET30a中,连接反应条件为:优化的SgSTPS1基因和pET30a质粒(摩尔比为3:1)、1x连接Buffer,T4DNA连接酶,混合后,15℃放置16h完成连接反应,获得重组质粒(优化的SgSTPS1基因插入到表达载体pET30a后的重组质粒)。然后将其转化到大肠杆菌Top10和大肠杆菌BL21(DE3)克隆菌株中。挑取抗生素(kan)筛选得到的阳性菌落,提取质粒,通过酶切法和测序确认最终表达载体的准确性,由此得到将优化的倍半萜合成酶基因SgSTPS1插入到pET30a表达载体的重组质粒成功转化至原核细胞的阳性菌,命名为Top10-SgSTPS1(2)和BL21-SgSTPS1。将构建好的BL21-SgSTPS1菌进行大量培养,首先挑取单克隆BL21-SgSTPS1,接种到4mL的LB培养基中(含50μg/mL的硫酸卡那霉素),37℃,200rpm,待培养至OD600为0.5-0.8,向试管培养液中加入终浓度0.1mM IPTG,之后分别置于15℃和37℃诱导表达,得到大量表达倍半萜合成酶SgSTPS1的菌液。
3、油楠倍半萜合成酶SgSTPS1的原核表达、蛋白纯化和蛋白质检
取诱导后大量表达倍半萜合成酶SgSTPS1的菌液,12000rpm离心5min,去除上清液,加入PBS液重悬沉淀,最后加入SDS-PAGE上样缓冲液于100℃下加热样品10min,然后离心取上清电泳。电泳前10min时,100V稳压电泳,待溴酚蓝指示剂进入分离胶后200V稳压电泳至溴酚蓝带迁移至离凝胶底部1cm,取出凝胶用考马斯亮兰染色液染色,随后转入脱色液中,脱色至背景清晰。结果见图3,图3的M为蛋白marker,Lane 0为对照,Lane 1为15℃诱导16h,Lane 2为37℃诱导16h,箭头为目的蛋白SgSTPS1。
按上述方法,放大培养3L的表达菌,生长至OD600=0.8时,加终浓度0.1mM IPTG,15℃诱导16h后收集菌体。全菌采用50mM Tris(pH8.0),300mM NaCl,50mM Imidazole含1%Triton X-100,1mM DTT,1mM PMSF超声裂解,同时以50mM Tris(pH8.0),300mM NaCl,50mMImidazole缓冲液平衡Ni-IDA亲和层析柱,之后用不同浓度咪唑的平衡缓冲液洗脱目标蛋白,并收集每个洗脱组分进行SDS-PAGE分析检测。分析结果见图4,图4的M为蛋白marker,Lane 1为全菌破菌离心后上清,Lane 2为上清同Ni-IDA孵育后流出液,Lane 3-4为100mMImidazole的洗脱组分,Lane 5-11为300mM Imidazole的洗脱组分,箭头为目的蛋白。
经Ni-IDA亲和层析纯化,收集纯度浓度较好的Lane 9-10,并将其透析到1×PBS,10%Glycerol,pH7.4透析结束后用0.22μm膜过滤,并分装冻于-80℃。用BSA做标准品,采用Bradford法测定蛋白浓度。油楠倍半萜合成酶SgSTPS1的蛋白Western-blot检测实验操作流程参考《蛋白质电泳实验技术》郭尧君著,结果见图5,Lane M1为SDS-PAGE蛋白Marker,Lane 1为BSA蛋白,Lane 2为SgSTPS1蛋白,Lane M2为Western-blot Marker,抗体为Anti-His。
4、β-石竹烯的生物合成
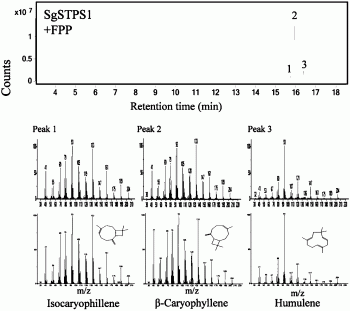
以法呢基焦磷酸(FPP)为底物,酶促反应体系为:25mM Tris-HCI(pH7.4),5mM二硫苏糖醇(DTT),100mM氯化钾,5mM氯化镁,10%甘油,底物法呢基焦磷酸(FPP)浓度为50μM,加入50μg纯化后的油楠倍半萜合成酶SgSTPS1蛋白,置于37℃反应1h。反应结束后,用固相微萃取SPME纤维PDMS 100μm顶空萃取挥发物质30min,250℃解吸3min进样。采用GC-MS联用仪(Agilent GC-MS 7890B-5977A)对催化产物进行检测。气相色谱柱为HP-5MS(30m×0.25mm),载气高纯He流速为1.0mL/min,升温程序为:50℃保持1min,以5℃/min速度升温至80℃,保持1min,再以10℃/min速度升温至220℃,保持10min,进样口温度为250℃,离子源EI 70eV温度为230℃,接口温度为250℃,采集质量范围为30-200amu,数据经NIST 14质谱谱库检索,并与标准谱图对照,鉴定各组分峰。样品总离子流图见图6,在保留时间为15.77、15.97和16.42处分别出现峰1、2和3,经NIST 14数据检索谱库检索比对,结果显示分别对应的化合物为异石竹烯(Isocaryophillene)、β-石竹烯(β-Caryophyllene)和α-石竹烯(Humulene),相似匹配度为分别为98、99和98,图中所示峰1为异石竹烯(Isocaryophillene),峰2为β-石竹烯(β-Caryophyllene),峰3为α-石竹烯(Humulene)。由此表明油楠倍半萜合成酶SgSTPS1可催化FPP合成β-石竹烯以及少量的异石竹烯和α-石竹烯。
序列表
<110> 中国林业科学研究院热带林业研究所
<120> 一种油楠倍半萜合成酶及其编码基因和应用
<160> 3
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1650
<212> DNA
<213> 油楠(Sindora glabra Merr.ex de Wit)
<400> 1
atggggcgac ccacggcaaa cttcagccca agtgtttggg gaaatcagtt cctttccatt 60
gcatcaggtc ccttgctgaa gaacaaagaa gcggaaattc atcaacattt actgaatctg 120
aaagaacaag tgaggaaaca actaaaaaat ggtgttgaag agccttctga gaagctaaac 180
ttgatcgata ccattcaacg gcttggagtg tctcaccatt ttgaaagaga gattgaagaa 240
tcattgaaac atctacataa gaatcctcct tcttcctgga atgccgaaga tatcgatgca 300
catcttttaa gcatttctct ctggtttcgg ttgcttagac aacaaggcta ttatgtctca 360
tgtgatgtat ttgataaatt caaagatgac aaaggcatgt tcaagacaac attgattgat 420
gatgtggaag gaatgttggc cttgtatgaa gctacatatc tcggcattcg tggggaagaa 480
attcttgatc aagtgctaga atttacaatg tttcatctta aatcaagatt ggaaggcatg 540
acaccctacc ttcgagaaag agcagatcgt gctttgtact gtcctataaa caaaggttta 600
ccacgaattg agacgcgata tttcatttcg atgtactcaa aaaaggattc tcgaaatgat 660
ctgttgctag aatttgccat gctagacttc aatatcctgc agcaacaata tcagaaggaa 720
ttaagccacc tatcagagtg gtacaaaaaa ttagattttg taagcaaggt tccttacacc 780
agagacagaa ttgttgaagg ctacttttgg cctttgggag catactttga gaatcaatat 840
aacaaaggaa gaataattgt aagcaaactg atattggttc ttacagcttt ggatgatacc 900
tatgatgcct acggtactgt agatgagctc aaactcttca ctgaagcaat taagagatgg 960
gatattaata tggtggcttc tctccctgaa tgcatgaaag tggtattcca agctatttta 1020
gatttgttgg atgaaatgga gttgctaacg gaagcggatg gaatatcatg ctttgttgaa 1080
tatgttaaac cagcgcttca agatttagca aaatcatact tgcttgaagc tgagtggagg 1140
gacaaaggct atataccaac atatgaggag tatatagcaa atggagtttt ttcttgtggt 1200
tacccagctg ttgaaatggc gtccttactt ggtcttggaa aaattgcaac caaagaagtg 1260
tttgattgga tttctaatgt acctaaaatt gttagagctt catcaattat gtgtagattg 1320
acagatgatt tggcttctca taagtttgaa caaaacagag aacatgttgg atctgcaatt 1380
gagtgttaca tgaagcaata tggggtttca gaagaagaag cctataaaat gctgctaaag 1440
gagatagaga atggatggaa ggatcttaac gaagagtaca tgaagccaaa tggtgttcca 1500
aaggtggtac ttaagtgtgt attgaatttt tcacgagtga ttgagtttct gtacggtcac 1560
tttgttgata aatacacaaa tgcagagatg ctgaaggatc acattgcttc actttttgtg 1620
gatcccattg ccattgatgt caacaaatag 1650
<210> 2
<211> 549
<212> PRT
<213> 油楠(Sindora glabra Merr.ex de Wit)
<400> 2
Met Gly Arg Pro Thr Ala Asn Phe Ser Pro Ser Val Trp Gly Asn Gln
1 5 1015
Phe Leu Ser Ile Ala Ser Gly Pro Leu Leu Lys Asn Lys Glu Ala Glu
202530
Ile His Gln His Leu Leu Asn Leu Lys Glu Gln Val Arg Lys Gln Leu
354045
Lys Asn Gly Val Glu Glu Pro Ser Glu Lys Leu Asn Leu Ile Asp Thr
505560
Ile Gln Arg Leu Gly Val Ser His His Phe Glu Arg Glu Ile Glu Glu
65707580
Ser Leu Lys His Leu His Lys Asn Pro Pro Ser Ser Trp Asn Ala Glu
859095
Asp Ile Asp Ala His Leu Leu Ser Ile Ser Leu Trp Phe Arg Leu Leu
100 105 110
Arg Gln Gln Gly Tyr Tyr Val Ser Cys Asp Val Phe Asp Lys Phe Lys
115 120 125
Asp Asp Lys Gly Met Phe Lys Thr Thr Leu Ile Asp Asp Val Glu Gly
130 135 140
Met Leu Ala Leu Tyr Glu Ala Thr Tyr Leu Gly Ile Arg Gly Glu Glu
145 150 155 160
Ile Leu Asp Gln Val Leu Glu Phe Thr Met Phe His Leu Lys Ser Arg
165 170 175
Leu Glu Gly Met Thr Pro Tyr Leu Arg Glu Arg Ala Asp Arg Ala Leu
180 185 190
Tyr Cys Pro Ile Asn Lys Gly Leu Pro Arg Ile Glu Thr Arg Tyr Phe
195 200 205
Ile Ser Met Tyr Ser Lys Lys Asp Ser Arg Asn Asp Leu Leu Leu Glu
210 215 220
Phe Ala Met Leu Asp Phe Asn Ile Leu Gln Gln Gln Tyr Gln Lys Glu
225 230 235 240
Leu Ser His Leu Ser Glu Trp Tyr Lys Lys Leu Asp Phe Val Ser Lys
245 250 255
Val Pro Tyr Thr Arg Asp Arg Ile Val Glu Gly Tyr Phe Trp Pro Leu
260 265 270
Gly Ala Tyr Phe Glu Asn Gln Tyr Asn Lys Gly Arg Ile Ile Val Ser
275 280 285
Lys Leu Ile Leu Val Leu Thr Ala Leu Asp Asp Thr Tyr Asp Ala Tyr
290 295 300
Gly Thr Val Asp Glu Leu Lys Leu Phe Thr Glu Ala Ile Lys Arg Trp
305 310 315 320
Asp Ile Asn Met Val Ala Ser Leu Pro Glu Cys Met Lys Val Val Phe
325 330 335
Gln Ala Ile Leu Asp Leu Leu Asp Glu Met Glu Leu Leu Thr Glu Ala
340 345 350
Asp Gly Ile Ser Cys Phe Val Glu Tyr Val Lys Pro Ala Leu Gln Asp
355 360 365
Leu Ala Lys Ser Tyr Leu Leu Glu Ala Glu Trp Arg Asp Lys Gly Tyr
370 375 380
Ile Pro Thr Tyr Glu Glu Tyr Ile Ala Asn Gly Val Phe Ser Cys Gly
385 390 395 400
Tyr Pro Ala Val Glu Met Ala Ser Leu Leu Gly Leu Gly Lys Ile Ala
405 410 415
Thr Lys Glu Val Phe Asp Trp Ile Ser Asn Val Pro Lys Ile Val Arg
420 425 430
Ala Ser Ser Ile Met Cys Arg Leu Thr Asp Asp Leu Ala Ser His Lys
435 440 445
Phe Glu Gln Asn Arg Glu His Val Gly Ser Ala Ile Glu Cys Tyr Met
450 455 460
Lys Gln Tyr Gly Val Ser Glu Glu Glu Ala Tyr Lys Met Leu Leu Lys
465 470 475 480
Glu Ile Glu Asn Gly Trp Lys Asp Leu Asn Glu Glu Tyr Met Lys Pro
485 490 495
Asn Gly Val Pro Lys Val Val Leu Lys Cys Val Leu Asn Phe Ser Arg
500 505 510
Val Ile Glu Phe Leu Tyr Gly His Phe Val Asp Lys Tyr Thr Asn Ala
515 520 525
Glu Met Leu Lys Asp His Ile Ala Ser Leu Phe Val Asp Pro Ile Ala
530 535 540
Ile Asp Val Asn Lys
545
<210> 3
<211> 1650
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
atgggtcgtc cgaccgcaaa ttttagtccg agcgtttggg gcaaccagtt tctgtccatt 60
gcaagcggtc cgctgctgaa aaacaaagaa gcggaaattc accagcatct gctgaatctg 120
aaagagcagg tccgcaaaca gctgaaaaac ggcgttgaag aaccgagcga aaaactgaac 180
ctgatcgata ccattcaacg tctgggcgtt agccaccatt tcgaacgcga aatcgaagag 240
agcctgaaac acctgcacaa aaatccgccg agtagctgga acgcagaaga tattgacgcg 300
catctgctgt ccatcagtct gtggtttcgt ctgctgcgtc aacagggtta ttacgttagc 360
tgcgacgtct tcgacaaatt caaagacgac aaaggcatgt tcaaaaccac cctgatcgac 420
gacgtcgaag gtatgttagc gctgtacgaa gcaacctatc tgggtattcg cggcgaagaa 480
atcctggatc aggttctgga gttcaccatg ttccacctga aaagccgtct ggaaggtatg 540
accccgtatt tacgcgaacg cgcagatcgc gcactgtatt gtccgatcaa caaaggcctg 600
ccgcgtattg aaacccgcta tttcatcagc atgtacagca aaaaagacag ccgcaacgac 660
ctgctgttag aattcgcgat gctggacttc aacatcctgc agcagcagta ccagaaagaa 720
ctgagccacc tgagcgagtg gtacaaaaaa ctggacttcg tcagcaaagt tccgtatacc 780
cgcgatcgca tcgttgaagg ttatttctgg ccgctgggcg catattttga aaaccagtac 840
aacaaaggcc gcatcatcgt cagcaaactg attctggttc tgaccgcgtt agacgatacc 900
tatgacgcat acggtaccgt tgacgaactg aaactgttca ccgaagcgat caaacgctgg 960
gacattaaca tggttgcctc tctgccggaa tgcatgaaag tcgttttcca ggcgatcctg 1020
gacttactgg acgaaatgga actgctgacc gaagctgacg gtattagctg cttcgtcgaa 1080
tatgttaaac cggcactgca ggatctggct aaaagctacc tgttagaagc ggagtggcgc 1140
gataaaggct atatcccgac ctacgaagaa tatatcgcga acggcgtttt tagctgcggt 1200
tatccggccg ttgaaatggc aagcttactg ggtctgggca aaattgcgac caaagaagtc 1260
ttcgactgga tcagcaacgt cccgaaaatc gttcgcgctt cctctattat gtgtcgtctg 1320
accgacgatc tggcgagcca taaattcgaa cagaatcgcg aacacgttgg ttccgctatt 1380
gagtgctaca tgaaacagta cggcgtcagc gaagaagaag cgtacaaaat gctgctgaaa 1440
gagatcgaga acggctggaa agacctgaac gaagaatata tgaaaccgaa cggggttccg 1500
aaagtcgttc tgaaatgcgt cctgaacttc agccgcgtta ttgaattcct gtacggccat 1560
ttcgtcgaca aatacaccaa cgcggagatg ctgaaagatc acatcgcgag cctgttcgtt 1620
gatccgattg ccatcgacgt caacaaataa 1650
一种油楠倍半萜合成酶及其编码基因和应用专利购买费用说明
![]()
Q:办理专利转让的流程及所需资料
A:专利权人变更需要办理著录项目变更手续,有代理机构的,变更手续应当由代理机构办理。
1:专利变更应当使用专利局统一制作的“著录项目变更申报书”提出。
2:按规定缴纳著录项目变更手续费。
3:同时提交相关证明文件原件。
4:专利权转移的,变更后的专利权人委托新专利代理机构的,应当提交变更后的全体专利申请人签字或者盖章的委托书。
Q:专利著录项目变更费用如何缴交
A:(1)直接到国家知识产权局受理大厅收费窗口缴纳,(2)通过代办处缴纳,(3)通过邮局或者银行汇款,更多缴纳方式
Q:专利转让变更,多久能出结果
A:著录项目变更请求书递交后,一般1-2个月左右就会收到通知,国家知识产权局会下达《转让手续合格通知书》。
动态评分
0.0