专利摘要
本发明公开了一种高效提高酶热动力学稳定性的通用策略;该策略的具体实施路线为:以酶活性部位的催化残基为中心,根据该酶晶体结构中的B‑factor值来选择周围以内的高柔性残基为靶标位点,通过突变筛选,获得酶热动力学稳定性提高的突变体。在该策略的指导下,显著提高了结构高度复杂的褶皱假丝酵母脂肪酶1的热稳定性。结合结构上最小蛋白(LipA)和中度复杂的蛋白(CalB)的稳定性变化结果,发现距离催化残基附近的高B‑factor残基的突可以显著提高酶突变的成功率。同时,借助突变体结构‑功能关系的系统分析将为深入研究酶区域稳定化规律、发展酶稳定化技术奠定了基础。
权利要求
1.一种提高酶热动力学稳定性的通用方法,其特征在于,以酶活性部位的催化残基为中心,根据该酶晶体结构中的B-factor值来选择距离催化残基 以内的高柔性残基为靶标位点,通过突变筛选,获得酶热动力学稳定性提高的突变体。
2.如权利要求1所述的提高酶热动力学稳定性的通用方法,其特征在于,所述筛选为三级筛选,包括荧光平板的初筛和两次96孔板的复筛。
3.如权利要求2所述的提高酶热动力学稳定性的通用方法,其特征在于,所述荧光平板的初筛包括突变体单克隆65℃热处理40min,室温冷却20min后在软胶培养基中30℃下培养5h,取出在紫外光下观察荧光,显示出较大的荧光圈即为高热稳定性和活性的候选菌株;所述两次96孔板的复筛包括:第一个96孔板的边沿和中间各取一个孔培养野生型LIP1作为对照,在PCR仪上58℃热处理15min之后,测定水解对硝基苯酚丁酸酯的活力,从失活率高于野生型20%的作为筛选标准,筛选到的克隆进入下一步96孔板筛选;每个阳性克隆转入一列4个孔中培养,分析热处理后的失活率;真正的阳性则表现出4个孔中的失活率都要比野生性要高,且提高幅度相近。
4.如权利要求1所述的提高酶热动力学稳定性的通用方法,其特征在于,所述方法还包括将突变筛选出的酶热动力学稳定性提高的突变体,通过有序叠加组合突变获得酶热动力学稳定性更高的突变体的步骤。
5.如权利要求4所述的提高酶热动力学稳定性的通用方法,其特征在于,所述有序叠加组合突变具体实施过程为:按照单点突变体稳定性提高幅度从大到小的顺序,逐级组合,直到所有突变位点都被引入到一个突变体中。
6.如权利要求1所述的提高酶热动力学稳定性的通用方法,其特征在于,所述酶选自LipA、Ca1B或具有如SEQ ID NO:1所示氨基酸序列的褶皱假丝酵母脂肪酶LIP1。
7.如权利要求6所述的提高酶热动力学稳定性的通用方法,其特征在于,选择距离LipA、Ca1B或LIP1的催化残基 和相对B-factor值在60-100的区域为突变热区。
8.如权利要求6所述的提高酶热动力学稳定性的通用方法,其特征在于,所述酶为Lip1时,以如SEQ ID NO:1所示序列为参考序列,选择到的突变热区对应的位点包括344、434、133、121。
9.如权利要求8所述的提高酶热动力学稳定性的通用方法,其特征在于,在位点344、434、133或121处发生单点突变;其中,344位点的苯丙氨酸突变为异亮氨酸或蛋氨酸,434、133和121位点的苯丙氨酸突变为络氨酸。
10.如权利要求8所述的提高酶热动力学稳定性的通用方法,其特征在于,通过位点344、434、133和121处发生单点突变进行有序叠加组合突变;其中,344位点的苯丙氨酸突变为异亮氨酸或蛋氨酸,434、133和121位点的苯丙氨酸均突变为络氨酸。
说明书
技术领域
本发明涉及一种高效提高酶热动力学稳定性的通用策略。通过Pymol和B-FITTER软件分析酶催化残基 以内高柔性的氨基酸;借助饱和突变库筛选出热动力学稳定性提高的突变体。具体涉及褶皱假丝酵母脂肪酶1的突变体,及突变体的结构与功能分析。
背景技术
酶是一种高效的生物催化剂,与化学催化剂相比,酶分子具有高效性、高专一性和反应条件温和无污染等优点,在科研和工业生产中有着重要的应用价值。然而在高压、高温、极端pH等恶劣条件下,酶容易解链而失去活性,这严重限制了酶的广泛应用,因此,如何提高酶的稳定性成为当前研究的一个重要课题。
随着结构生物学的发展,越来越多的酶结构得到了解析。目前已有多篇基于单酶结构分析,进行理性设计提高酶稳定性的报道。如Cerdobbel(Cerdobbel,A.et al.Increasing the thermostability of sucrose phosphorylase by a combination of sequence and structure based mutagenesis[J].Protein Engineering Design and Selection,2011,24(11):829-834.)等人通过突变氨基酸产生静电相互作用,提高了蔗糖磷酸化酶60℃的半衰期2.6倍;Wang(Wang.Y.et al.Improved thermal performance of Thermomyces lanuginosus GH11xylanase by engineering of an N-terminal disulfide bridge[J].Bioresource Technology,2012,112:275-279.)等人通过引入二硫键使得嗜热真菌木聚糖酶在70℃的半衰期提高20倍;Gallardo(Gallardo, et al.Structural insights into the specificity of xynl0B from Paenibacillus barcinonensis and its improved stability by torced protein evolution[J].Journal of Biological Chemistry,2010,285(4):2721-2733.)等人通过增加蛋白质的疏水相互作用,提高了类芽孢杆菌木聚糖酶Xyn10B在50℃的半衰期20倍。这些成果多为个例分析,尚无规律可循。随着蛋白质组学概念的引入,酶稳定化策略有了新的发展。通过对不同来源的植酸酶多序列分析,找到其中一致性位点指导突变,成功将其Tm值提升22℃(Lehmann,M.et al.From DNA sequence to improved functionality:Using protein sequence comparisons to rapidly design a thermostable consensus phytase[J].Protein Engineering,2000,13(1):49-57.);对多个内酰胺酶序列进行生物统计学分析,推测其祖先基因,成功将其Tm值提升7℃(Bershtein,S.et al.Intense neutral drifts yield robust and evolvable consensus proteins[J].Journal of Molecular Biology,2008,379(5):1029-1044.);利用结构数据库指导的酶重组技术,成功将纤维素酶在63℃下的半衰期提高30倍(Heinzelman,et al.A family of thermostable fungal cellulases created by structure-guided recombination[J].Proceedings of the National Academy of Sciences,2009,106(14):5610-5615.)。对酶系的系统分析,有效提升酶稳定性改造的效率。但是,这种方法需要大量的数据进行统计分析,数据量的不足也限制了该方法的准确性与应用范围。因此,是否能够专注与酶稳定性相关的关键部位,建立简单易行,快速高效,通用性强的酶稳定化新策略,是本发明关注的重点。
为了深入了解酶的稳定化机制,学者们比较了嗜热酶与中温酶的结构,发现前者在某些区域上具有更强的刚性,从而带来了酶稳定区域化研究的高潮,尤其是酶结构上的柔性区域。在蛋白质晶体结构中,B-factor成为识别柔性氨基酸的重要指标。B-factor是指酶蛋白质中每个组成氨基酸原子的电子密度在平衡位点的空间“模糊度”,即原子是在振动当中,B-factor反应出原子的振动幅度,从而转化为氨基酸的柔性;B-factor越大,则氨基酸柔性越大。根据这个原理,德国知名学者Reetz(Reetz,M.T.et al.Iterative saturation mutagenesis on the basis of B-factors as a strategy for increasing protein thermostability[J].Angewandte Chemie International Edition,2006,45(46):7745-7751.)以自然界中最小的枯草芽孢杆菌脂肪酶(LipA:181个氨基酸)为研究对象,选取了整个酶分子中B-factor最大的10个残基作为靶标位点,构建迭代饱和突变库筛选稳定性提高的突变体。这10个靶标位点全部位于酶分子表面。经过多轮的筛选,最优突变体在55℃的半衰期从2min提升至16h,结果十分显著。该文献报道后,得到了学术界广泛的关注,现已有多篇文献利用B-factor分析对酶稳定性进行改造。但是,当研究对象为链霉菌磷脂酶(PLD:509个氨基酸)( J.et al.Improving thermostability of phosphatidylinositol synthesizing Streptomyces phospholipase D[J].Protein Engineering Design and Selection,2012,25(8):415-424.)时,对酶B-factor最高的残基进行突变,酶稳定性提升幅度并不明显,在65℃的半衰期由25.9min提升至34.6min。从而说明表面区域的柔性改造对于分子更为复杂的蛋白仍存在缺陷。
Candida rugosa lipasel(LIP1)具有较大的分子量和复杂的结构(534个氨基酸组成)。而且,LIP1又是一种重要的工业应用性脂肪酶,有着高的催化效率和宽范围的底物选择性(Akoh,C.C.et al.Protein engineering and applications of Candida rugosa lipase isoforms[J].Lipids,2004,39(6):513-526.);在医药应用领域,它的手性常用于拆分重要的医药产品,如:萘普生、酮洛芬和布洛芬等(Chavez-Flores,et al.Facile conversion of racemic ibuprofen to(s)-ibuprofen[J].Tetrahedron:Asymmetry,2012,23(3):237-239;Lee,E.G.,et al.Enantioselective hydrolysis of racemic naproxen methyl ester by two-step acetone treated Candida rugosa lipase[J].Process Biochemistry,2001,37(3):293-298;Rangheard,M.S.,et al.Multi-competitive enzymatic reactions in organic media:A simple test for the determination of lipase fatty acid specificity[J].Biochimica et Biophysica Acta(BBA)-Lipids and Lipid Metabolism,1989,1004(1):20-28;Tsai,S.W.et al.Surfactant enhancement of(s)-naproxen ester productivity from racemic naproxen by lipase in isooctane[J].Biotechnology and Bioengineering,1996,51(2):148-156;López,N.et al.Reactivity of pure Candida rugosa lipase isoenzymes(LIP1,LIP2,and LIP3)in aqueous and organic media.Influence of the isoenzymatic profile on the lipase performance in organic media[J].Biotechnology Progress,2004,20(1):65-73.)。但是LIP1弱稳定性限制了它在高温、高压等工业转化条件下的应用(Chang,S.W.et al.Codon optimization of Candida rugosa LIP1 gene for improving expression in Pichia pastoris and biochemical characterization of the purified recombinant LIP1 lipase[J].Journal of Agricultural and Food Chemistry,2006,54(3):815-822.)。稳定性差的LIP1容易失活变性,失去催化反应功能。因此,提高LIP1的稳定性在工业应用上也有着重大意义。
高稳定性的酶不仅在高温环境下表现出高效的催化活力,同时对反应器冷却系统要求较低,减少能耗;还可以提高反应产物的纯度,减少细菌对转化产物的污染等优点。目前,对于LIP1热稳定性研究主要来自于不同载体制备的固定化酶(Knezevic,Z.et al.Immobilization of lipase from Candida rugosa on C supports by covalent attachment[J].Biochemical Engineering Journal,2006,30(3):269-278; G.,Kaya,B.,Ar1ca,M.Y.Immobilization of Candida rugosa lipase onto spacer-arm attached poly(GMA-HEMA-EGDMA)microspheres[J].Food Chemistry,2005,92(2):261-268.)。对LIP1热稳定性改造也将进一步揭示出它在结构与功能上的关系。
发明内容
本发明目的在于克服上述现有技术存在的不足,提供一种高效提高酶热动力学稳定性的通用策略(方法);显著提高了结构上高度复杂蛋白LIP1的热动力学稳定性,并进一步解释酶活性中心结构与功能的关系。具体而言,本发明以LIP1的催化残基Ser209为中心,选择了周围 以内18个具有最高B-factor的残基进行饱和突变。在三级筛选方法的分析下,获得了五个单点热稳定性提高的突变体,且活力也有不同程度提升。借助于有序叠加组合突变,快速实现了单点突变的协同效应。
本发明的目的是通过以下技术方案来实现的:
本发明涉及一种提高酶热动力学稳定性的通用方法,以酶活性部位的催化残基为中心,根据该酶晶体结构中的B-factor值来选择周围 以内的高柔性残基为靶标位点,通过突变筛选,获得酶热动力学稳定性提高的突变体。该活性中心是指酶催化残基附近的结构区域,区别于复杂结构的表面残基。而残基柔性的大小主要是根据晶体结构中的B-factor值来判断;B-factor越大,则柔性越高;B-factor越小,刚性越强。
优选的,所述筛选为三级筛选,包括荧光平板的初筛和两次96孔板的复筛。
优选的,所述荧光平板的初筛包括突变体单克隆65℃热处理40min,室温冷却20min后在软胶培养基中30℃下培养5h,取出在紫外光下观察荧光,显示出较大的荧光圈即为高热稳定性和活性的候选菌株;所述两次96孔板的复筛包括:第一个96孔板的边沿和中间各取一个孔培养野生型LIP1作为对照,在PCR仪上58℃热处理15min之后,测定水解对硝基苯酚丁酸酯(pNP-C4)的活力,从失活率高于野生型20%的作为筛选标准,筛选到的克隆进入下一步96孔板筛选;每个阳性克隆转入一列4个孔中培养,分析热处理后的失活率;真正的阳性则表现出4个孔中的失活率都要比野生性要高,且提高幅度相近。
优选的,所述方法还包括将突变筛选出的酶热动力学稳定性提高的突变体,通过有序叠加组合突变获得酶热动力学稳定性更高的突变体的步骤。
优选的,所述有序叠加组合突变具体实施过程为:按照单点突变体稳定性提高幅度从大到小的顺序,逐级组合,直到所有突变位点都被引入到一个突变体中。本发明中,有序叠加组合突变方案通过用稳定性提高幅度最大与次大的单点突变逐级叠加组合,快速获得了稳定性提高最显著的突变体。
优选的,所述酶选自CalB、LipA或具有如SEQ ID NO:1所示氨基酸序列的褶皱假丝酵母脂肪酶LIP1。该LIP1酶对应的核苷酸序列如SEQ ID NO:9所示。
优选的,选择距离LipA、CalB或LIP1的催化残基 和相对B-factor值在60-100的区域为突变热区。本发明从LipA、CalB和LIP1三种不同结构复杂度的突变结果中发新的规律:距离催化残基 和相对B-factor值在60-100的区域为突变热区,能够提高突变效率。其中,相对B-factor是指:催化残基 以内,最高B-factor值为对照,其余残基的B-factor与其比较的百分数。
优选的,以LIP1的催化残基Ser209为中心,选择了距离Ser209 附近的具有最高B-factor值的残基为突变热区,进行饱和突变。本发明在距离催化残基 附近的高B-factor残基(B-factor相对值在60-100)为突变热区,可以提高酶突变的成功率。优选的,所述酶选择LIP1时,以如SEQ ID NO:1所示序列为参考序列,选择到的突变热区对应的位点包括344、434、133、121。
优选的,在位点344、434、133或121处发生单点突变;其中,344位点的苯丙氨酸突变为异亮氨酸或蛋氨酸,434、133和121位点的苯丙氨酸突变为络氨酸。对应的LIP1的突变体分别是Phe344Ile(F344I)、Phe344Met(F344M)、Phe434Tyr(F434Y)、Phe133Tyr(F133Y)、Phe121Tyr(F121Y),对应的的氨基酸序列依次如SEQ ID NO:2~SEQ ID NO:6所示,对应的核苷酸序列依次如SEQ ID NO:10~SEQ ID NO:14所示。
上述对344位点进行突变采用的引物序列如SEQ ID NO:17、SEQ ID NO:18所示;对133位点进行突变采用的引物序列如SEQ IDNO:21、SEQ IDNO:22所示;对121位点进行突变采用的引物序列如SEQ ID NO:27、SEQ ID NO:28所示;对434位点进行突变采用的引物序列如SEQ ID NO:37、SEQ ID NO:38所示。
优选的,通过位点344、434、133和121处同时发生单点突变进行有序叠加组合突变;其中,344位点的苯丙氨酸突变为异亮氨酸或蛋氨酸,434、133和121位点的苯丙氨酸均突变为络氨酸。对应的LIP1的突变体分别是Phe344Ile/Phe434Tyr/Phe133Tyr/Phe121Tyr(F344I/F434Y/F133Y/F121Y)、Phe344Met/Phe434Tyr/Phe133Tyr/Phe121Tyr(F344M/F434Y/F133Y/F121Y),对应的的氨基酸序列依次如SEQ ID NO:7、SEQ ID NO:8所示,对应的核苷酸序列依次如SEQ ID NO:15、SEQ ID NO:16所示。本发明通过有序叠加组合突变方案,获得热稳定性提高幅度最大的两个突变体VarA3(F344M/F434Y/F133Y/F121Y)和VarB3(F344I/F434Y/F133Y/F121Y)。
本发明的原理在于:
对于结构复杂的蛋白质,将B-factor与其他理性设计思路相结合,可能会更好地指导酶稳定性改造。
活性中心是酶的结构中最为关键部位,局部构象的适度柔性有助于更好的结合底物,促进酶的高效催化性。然而酶的活性中心在整个结构中具有一定的脆弱性,大量不同类型酶的变性结果显示出,酶的失活往往发生在可检测的蛋白质整体构象变化之前,即“先失活,后变性”。另外,发明人所在实验室前期对脂肪酶CalB的研究中,发现酶活性中心区域的突变除了影响酶催化之外,还可以显著改善酶的稳定性。因此,本发明提出了“酶活性中心稳定化”策略来改造酶的稳定性,特别针对于那些结构上更为复杂酶的稳定性改造,有着重要的指导意义。
“酶活性中心稳定化”策略的操作过程是:以酶的催化残基为中心,通过Pymol和B-FITTER软件选取附近 以内高柔性氨基酸为靶标。实行定点饱和突变构建突变库,利用合适的筛选方法获得稳定性提高的突变体。
与现有技术相比,本发明具有如下有益效果:
1、本发明在“酶活性中心稳定化”策略的指导下,通过蛋白质工程技术,成功获得热稳定性显著提高的突变体Phe344Ile、Phe344Met、Phe434Tyr、Phe133Tyr、Phe121Tyr。其中Phe344Ile和Phe344Met变化最大,它们的 比野生型提高了7.7℃和7.9℃,同时水解对硝基苯酚辛酸酯(pNP-C8)的催化效率(kcat/Km)分别为野生型的0.94和1.23倍,即酶稳定性提高的同时催化效率没有受到太大影响。
2、通过有序叠加组合突变,快速获得了稳定性进一步提高的突变体VarB3,最适温度提高了15℃, 值比野生型也提高了13℃,半衰期提高了约40倍,热力学稳定性的Tm值提高13℃,催化效率与野生型相同。
3、本发明同时分析了三个典型α/β水解酶类的脂肪酶(LipA、CalB和LIP1),它们在分子量具有从小到大,结构上具有从简单到复杂的特征;结合它们在活性中心高柔性残基的突变显著提高酶的稳定性,预示着“酶活性中心稳定化”策略的通用性;根据这三种典型酶的突变结果,统计分析出:距离催化残基 附近的高B-factor残基(B-factor相对值在60-100)为突变热区,可以提高酶突变的成功率。这为其它酶类的改造带来重要的理论指导。
附图说明
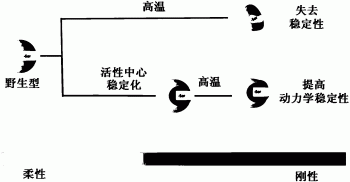
图1为酶活性中心稳定化策略的示意图;
图2为活性中心所选取的靶标位点示意图,其中,A为LIP1的“盖子”打开结构图,B为LIP1的“盖子”关闭结构图;
图3为高热稳定性和高催化活力突变体的三级筛选方案示意图;
图4为有序叠加组合的方案流程图;
图5为脂肪酶LIP1和突变体的底物选择性示意图;
图6为脂肪酶LIP1和突变体的热力学稳定性示意图;
图7为野生型LIP1和突变体VarB3内部的分子作用力分析图,其中,A为突变位点121和133周围区域的结构变化,B为突变位点344周围区域的结构变化,C为突变位点434周围区域的结构变化;
图8为不同分子量脂肪酶的拓扑结构图,其中,A为最小结构酶LipA,B为中度复杂结构酶CalB,C为结构高度复杂的酶LIP1;
图9为活性中心热点氨基酸的空间分布。
具体实施方式
本发明涉及一种通用且高效提高酶热动力学稳定性的策略。该策略的具体实施路线为:以酶活性部位的催化残基为中心,根据该酶晶体结构中的B-factor值来选择周围 以内的高柔性残基(高B-factor)为靶标位点,通过构建饱和突变库,筛选到热动力学提高的突变体(见图1)。在该策略的指导下,以催化残基Ser209为中心,显著提高了结构高度复杂的褶皱假丝酵母脂肪酶1(Candida rugosa lipase1:LIP1)的热稳定性。结合结构上最小蛋白(LipA)和中度复杂的蛋白(CalB)的稳定性变化结果,发现距离催化残基 附近的高B-factor残基(B-factor相对值在60-100)为突变热区,可以提高酶突变的成功率。同时,借助突变体结构-功能关系的系统分析将为深入研究酶区域稳定化规律、发展酶稳定化技术奠定了基础。
下面结合附图和具体实施方式对发明做进一步说明:
实施例1、突变位点的选择
本发明所涉及的Candida rugosa的脂肪酶1的野生型(WT)氨基酸序列为SEQ ID NO:1所示,其核苷酸序列为SEQ ID NO:9所示。
在PDB数据库中搜索LIP1的晶体结构,选择分辨率最高的结构作为研究对象。由于LIP1在活性口袋上方有一个称为“盖子”的螺旋片段,导致LIP1在溶液中具有两种结构:Open和Closed,因此在选择靶标位点时分别对两种结构同时进行了分析。分别都以催化残基Ser209为中心,分析周围 以内的所有氨基酸(用PyMOL命令:PyMOL->select AA,polymer within 10 of Ser209来查找)。再使用B-FITTER软件分析这些氨基酸的B-factor值,按照值从大到小的顺序排列,选取每个结构中的前11个最大B-factor氨基酸作为靶标位点(见表1)。在这些残基中有3个出现重复选取,故最终对两种结构总共选取了18个残基作为靶标位点(见图2)。下面是这些靶标位点在结构中的具体位置:Phe344和Phe345都位于α12螺旋上;Phe434位于β13折叠;Ser84、Lys85、Phe87和Glu88都位于α1和β5之间的loop上;Phe121、Gly122、Gly123、Gly124、Phe125、Glu126、Val127和Phe133都位于β6和α3之间的loop上;Phe296和Leu302位于α10和α11之间的loop上;Gly414位于α15和α16之间的拐角处。
表1活性中心靶标位点的B-factor值和到催化残基Ser209的距离
为了更加有效的进行饱和突变,把相邻位点的氨基酸组合在一起构建突变库,结果构建了以下12个突变库:LibraryA(Phe344,Phe345)、Library B(Leu302)、Library C(Phe133)、Library D(Gly124,Phe125)、Library E(Glu126,Val127)、Library F(Phe121)、LibraryG(Phe87,Glu88)、LibraryH(Gly122,Gly123)、LibraryI(Ser84,Lys85)、Library J(Phe296)、Library K(Phe434)、Library L(Gly414)。所有突变库以球棍模型显示在图2中。
实施例2、定点饱和突变构建基因突变库
构建饱和突变库的引物见表2,
表2用于构建饱和突变库的引物
在PrimerSTAR max DNA聚合酶(TaKaRa公司)的作用下,以LIP1的野生型基因为模板,全质粒扩增。PCR反应条件:98℃预变性5min,每个循环98℃变性10s,55℃退火5s,72℃延伸2.5min,共30个循环;最后72℃延伸5min。将PCR产物纯化后直接转入大肠杆菌DH5α感受态,42℃热击90s,37℃孵育1h。之后将其涂布与含有25μg/ml Zeocin抗性的LB平板上,待克隆长出后,直接用无菌水将所有克隆从平板上洗脱下来,收集到一个试管里,用试剂盒提取混合质粒。用限制性酶切酶AvrII对提取到的混合质粒进行线性化,产物用试剂盒纯化后直接电击转入毕赤酵母GS115感受态细胞,在含有25μg/ml Zeocin抗性的YPD平板上30℃培养3d,得到含有突变基因的菌株。
实施例3、毕赤酵母感受态细胞的制备
从YPD平板上挑取毕赤酵母单菌落,接种于4ml YPD液体培养基中,30℃下振荡培养过夜,约24-48h左右,OD约6-8。将培养液分装到1.5ml EP管中,4℃、4000rpm、离心5min,弃上清。沉淀用冰冷的无菌水洗涤,4℃、4000rpm、离心5min,弃上清。沉淀用1ml醋酸锂溶液处理,沉淀重悬后,放置30℃下30min。之后4℃、4000rpm、离心5min,弃上清。(醋酸锂溶液:10mM pH7.5Tris-HCL;10mM DTT;100mM LiAC)沉淀用冰冷的无菌水洗涤,4℃、4000rpm、离心5min,弃上清。沉淀用冰冷的1M的D-山梨醇洗涤,4℃、4000rpm、离心5min,弃上清。彻底倒出上清,再加入1M的D-山梨醇50μl,制备成感受态细胞,准备下一步做转化。
(注意:感受态要现做现用,处理液中的DTT是在使用前加入,其它配好后于4℃保存,DTT单独于-20℃保存,可配成100倍的贮备液。)
实施例4、三级筛选饱和突变库
三级筛选方法如图3所示,主要包括荧光平板的初筛和两次96孔板的复筛;荧光平板初筛的方法:将YPD_Zeo抗性平板上长出的单克隆,通过影印纤维素膜转移到两个新的平板上;30℃培养两天,一个平板保存于冰箱,另一平板转入65℃的培养箱中;热处理40min,取出后室温冷却20min,将溶解的软胶培养基10ml,倒入该平板上;再次30℃下培养5h,在紫外光下观察荧光,高热稳定性和活性的突变体显示出较大的荧光圈。通过荧光圈筛选可以有效的淘汰在活性和稳定性上出现负突变的克隆。接着利用96孔板复筛,每个96孔的边沿和中间各取一个孔培养野生型LIP1作为对照,用YPD液体培养基在30℃,220rpm下培养72h。4000rpm离心96孔板30min,转培养上清液到另一孔板中,用无菌水将每个空中样品稀释4倍。稀释后的酶液分成两部分,一部分直接取10μl在酶标仪上测定水解pNP-C4的活力,保存数据,作为热失活前的活力。另一部分取50μl于96孔PCR板上,在PCR仪上58℃热处理15min。之后离心除去沉淀,取离心上清在酶标仪上测定残余活力,作为热失活后数据;将两次数据相比,计算失活率,从失活率高于野生型20%的作为筛选标准,筛选到的克隆进入下一步96孔筛选。为了排除初筛中出现的假阳性,将得到的单菌落转移到下一个新的96深孔板中,每个阳性克隆转入一列4个孔中培养,分析热处理后的失活率;真正的阳性则表现出4个孔中的失活率都要比野生性要高,且提高幅度相近。
实施例5、有序叠加组合突变
为了快速获得有益突变菌株的累积效应,采取了有序叠加组合突变方案,每次用最好的突变体组合次好的突变体。这个组合突变方案能够更加快速的得到组合突变结果,具体组合过程见图4。由于位点344处的两个单点突变体在所有突变体中具有最高幅度的提高,所以分别设计了两条有序叠加组合路径:从F344M到F121Y和F344I到F121Y。这两条路径除了在出发菌株不同外(分别是F344M和F344I),其余组合过程完全一样。两条途径中的组合突变体分别用新的名字代替:
VarA1(F344M/F434Y)、VarA2(F344M/F434Y/F133Y)、VarA3(F344M/F434Y/F133Y/F121Y)、VarB1(F344I/F434Y)、VarB2(F344I/F434Y/F133Y)、VarB3(F344I/F434Y/F133Y/F121Y)。从图4的组合结果显示出,每次的组合都能使得稳定性有不同程度提高。这些结果充分说明四个单点突变体之间存在着累积效应,而且证实了有序叠加组合突变方案是一种快速获得更好结果的有效途径。
实施例6、褶皱假丝酵母脂肪酶1野生型及突变体的表达和纯化
从YPD_Zeo平板上挑取单菌落于4ml的YPD培养基中培养,当OD600达到2时,按1%的接种量转入新的200ml YPD中表达,每12h取样检测活力,直到培养结束。
由于目的基因的N端设计了6×His的标签,可以直接利用Ni-亲和层析的方法纯化。LIP1在毕赤酵母中是以分泌性表达,最终表达产物主要在培养液中,故需要进行培养液的浓缩收集。因此,整个LIP1的纯化分为超滤浓缩和Ni-亲和层析两个步骤:
(一)超滤浓缩
酵母表达培养液在8000rpm的转速下离心20min,收集上清液;通过0.22μm的纤维膜过滤之后,转入Millipore的超滤装置中;用10kD的超滤膜包进行脱盐、脱色和浓缩,在浓缩过程中不断用加入ddH2O,直到浓缩液接近无色;用20mM的Tris-HCl 200ml再次将蛋白浓缩在100ml左右,转入镍柱亲和层析,并取样进行电泳鉴定和浓度测定。
(二)镍柱亲和层析
将水用0.22μm的滤膜过滤后,直接加5ml到镍柱上方→流完后加入5ml strip buffer(100mM EDTA,0.5M NaCl,50mM Tris-HCl,pH 7.5),将Ni2+和杂蛋白从载体NTA琼脂糖上洗涤下来→柱子加5ml水冲干→流完后加入5ml charge buffer(100mM NiSO4)→流完后加水冲洗→加入10ml binding buffer(50mM Tris-HCl,pH 7.5,20mM咪唑,0.5M NaCl)→流完后加入超滤浓缩后的酶液→流完后加入10ml binding buffer→流完后加入5ml wash buffer(200mM咪唑,0.5M NaCl,50mM Tris-HCl,pH 7.5)梯度洗脱,收集流出液→柱子加水冲洗,用20%乙醇保存在4℃冰箱中。
将洗脱液收集在一起转入透析袋,于50mM Tris-HCl,pH 7.5缓冲液中4℃透析24h,中间换两次透析液。最后收集蛋白,保存与-80℃冰箱备用。
用上述方法分别获得LIP1的突变体Phe344Ile(F344I)、Phe344Met(F344M)、Phe434Tyr(F434Y)、Phe133Tyr(F133Y)、Phe121Tyr(F121Y)、Phe344Ile/Phe434Tyr/Phe133Tyr/Phe121Tyr(F344I/F434Y/F133Y/F121Y)、Phe344Met/Phe434Tyr/Phe133Tyr/Phe121Tyr(F344M/F434Y/F133Y/F121Y)的突变体。并经过测序,上述突变体酶的氨基酸序列依次如SEQ ID NO:2~SEQ ID NO:8所示;对应的核苷酸序列依次如SEQ ID NO:10~SEQ ID NO:16所示。
实施例7、褶皱假丝酵母脂肪酶活力测定和底物选择性分析
用对硝基苯分辛酸酯(pNP-C8)作为底物,用乙腈配制成10mM的浓度。以50mM的pH 8.0Tris-HCl作为缓冲液,并含有1%(w/v)阿拉伯胶和2%(w/v)的脱氧胆酸钠(作为乳化剂)。在40℃水浴中,利用紫外分光光度计的测定单位时间内光的吸收值。具体反应体系为:取20μl的底物溶解在970μl的缓冲液中,40℃预热2min,加入10μl酶液,用移液枪吹匀反应体系,开始记录反应过程,大约2min后停止反应,按照下面公式(公式1)计算出酶的活力。1个酶活力单位(U)为:每分钟催化水解1μmol底物所需要的酶量。
A(U/mg):酶的比活力;ΔE(min-1):反应时间内光吸收405nm的变化值;vr(1):反应体积;ε(M-1·em-1):对硝基苯酚的摩尔吸光系数(1.6×104);d(cm):光线穿过的路径长度;ve(1):反应总体积;ce(mg/l):酶的浓度;实验数据为三次取平均值。
对于测定不同链长的pNP底物的活力,方法与上述相同,结果见图5。
实施例8、褶皱假丝酵母脂肪酶1及突变体的热稳定性和动力学参数分析
为了分析突变体的热稳定性变化,本发明利用终浓度0.2mM对硝基苯酚辛酸酯,分别对热稳定性常用参数t1/2(半衰期)、Topt(最适温度)和 ( 酶在不同温度下,热处理15min,活力失去一半时所对应的温度)进行了测定,测定酶活力的方法如实施例6。
在30~65℃的温度范围内,分析不同温度下pNP-C8的水解反应速率测出LIP1的最适温度。热稳定性检测通过将纯化好的LIP1(0.1mg/ml)每50μl分装在多个离心管中,放置于60℃下保温,在不同的时间间隔内取样,测定残余活力。根据公式2,可以换算出该温度下的半衰期。
t1/2=ln2/kd(公式2)
热稳定性参数 的测定方法:通过将纯化好的LIP1(0.1mg/ml)每50μl分装到96孔PCR板中,设置温度梯度40~80℃,对酶进行15min热失活处理;结束后4℃冷却10min,室温静置20min,4000rpm离心96孔PCR板30min,除去沉淀;分析上清液的残余活力,以相对残余活力和温度做出曲线图;根据波尔兹曼模型(公式3),在Origin8.0软件辅助下拟合,计算出 值。
y=A2+(A1-A2)/(1+exp((x-x0)/dx))(公式3)
动力学参数的分析是在pH 8.0,温度40℃的条件下,以pNP-C8为底物,分别测定底物浓度为4、10、15、20、50、100、150和200μM所对应的水解活力。利用LIP1对不同底物浓度的催化活力数据,借助软件Origin 8.0和米氏方程(公式4)拟合出函数曲线,从而得到Km和Vmax,再根据最大速率与酶浓度的函数方程(公式5),计算得到动力学常数Km,kcat和kcat/Km。
V=Vmax[S]/(Km+[S])(公式4)
Vmax=kcat×[E](公式5)
经过以上的测定方法,获得LIP1和突变体的动力学参数以及热稳定性参数见表3。
表3脂肪酶LIP1和突变体的动力学参数和热稳定性
实施例9、分析LIP1和突变体的热力学稳定性变化(Tm)
设定扫描温度范围为30~90℃,扫描速度为1℃/min。蛋白样品的浓度控制在0.5mg/ml,溶解在10mM的磷酸缓冲液中;开始扫描前先在30℃预平衡15min,Tm为最高波峰处对应的温度。
图6分析结果显示出所有突变体的Tm值相对于野生型都有了不同程度的提高,最小幅度的提升(ΔTm)也有4.5℃(F434Y,57.9℃)。值得注意的是,Tm值提高最大的并不是动力学稳定性提高最显著的Var3(ΔTm=12.7℃),而是三个位点组合体组合突变体VarA2(F344M/F434Y/F133Y),它的Tm值由野生型的53.4℃提升到70.8℃,提高了将近17.5℃。这个结果表明,动力学稳定性和热力学稳定性并非完全对应成正比例关系。
实施例10、分析稳定性突变体VarB3稳定性提高的机制
通过同源建模显示出突变体VarB3和野生型LIP1在结构上非常相似,为了合理解释导致蛋白稳定性提高的机制,需要对突变点附近Normalized B-factor降低的残基进行分子内部作用力的深入洞察。利用PyMOL软件分析了突变体VarB3模型结构和LIP1晶体结构(1CRL)的分子内部相互作用,详细结果见下图7。
从图7A上可以看出,位点121和133被突变成络氨酸,导致了7个新的氢键产生,其中突变后的Tyr133与水分子Wa、水分子Wb和Gly122形成了3个氢键,这3个氢键与突变前存在于水分子Wa、Wb和Gly122之间的4个氢键,总共7个氢键在Tyr133、Wa、Wb和Gly122之间形成了一个相互作用的氢键网络。而突变后的Tyrl21与水分子Wc、Phe128、Asn155和Tyr156形成了4个氢键,这4个氢键与突变前存在于水分子Wc和Phe128、Asn155之间的2个氢键,总共6个氢键在Tyr121、Wc、Phe128、Asn155和Tyr156之间也出现了一个相互作用的氢键网络。氢键网络是提高蛋白质稳定性的一个重要因素,这两个氢键网络的出现将有助于稳定残基120与136之间loop区域的摆动,从而也贡献了整体蛋白的稳定。
从图7B上可以明显发现位点344被突变成Ile后,α12螺旋明显延长。这主要是Ile的引入导致Phe345和Thr343的侧链发生了轻度偏转,拉近了Thr343与Gly346之间的距离,在它们之间产生了一个 新的氢键;同时也导致Phe345与Ser348之间的氢键键长由原来的 变为 Gly346在新形成氢键的拉动下使得它的侧链更靠近了Ser349,结果在Gly346和Ser349之间的两个氢键键长都由原来的 变为 所有这些更紧密的堆叠使得原来位于Thr347和Ser349之间的loop转变成了α螺旋,相应的延长了螺旋α12;loop转化为螺旋使得活性中心局部的刚性增强。
从图7C上发现,在位点434处苯丙氨酸突变成络氨酸形成了一个 的氢键,这个氢键出现在Tyr434与水分子Wd之间。仔细观察β13折叠会发现,在它上面的残基与周围氨基酸相互之间的氢键键长都发生了不同程度的减小,它们分别是:Tyr432与Asp479之间的氢键键长由原来的 变为 Ser433与Ile335之间的两个氢键键长分别由 和 变成了 和 Ser433与Met503之间的氢键键长由 变为 Leu435与Ile505之间的氢键键长由 变为 尽管这些氢键键长的变化较小,但多个微小变化的聚合将会带来β13折叠的刚性增强。从以上对结构详细的分析之后,发现氢键相互作用网络和活性中心密集的堆叠是导致局部刚性增强的主要原因,进而在整体上反应出突变体VarB3的稳定性提高。
实施例11、活性中心稳定化策略的通用性
为了探索酶活性中心对稳定性有重要影响的氨基酸分布特征,对不同结构复杂度酶蛋白的稳定性提高结果进行了详细分析,尤其是德国学者Reetz的研究成果(Reetz,M.T.et al.Iterative saturation mutagenesis on the basis of B-factors as a strategy for increasing protein thermostability[J].Angewandte Chemie International Edition,2006,45(46):7745-7751.)。结果发现Reetz突变的蛋白是“酶活性中心稳定化”策略的一个特例。LipA是一个最小的α/β水解酶折叠蛋白,它仅有181个氨基酸组成,从图8A中可以看出,结构上它仅有标准的α螺旋和β折叠形成活性部位,没有多余的二级结构包裹在蛋白质表面,LipA在结构上也具备了最简单特征。本发明选定的催化活性残基周围 以内的高柔性残基,对于LipA来说,几乎将整个蛋白都包含了进去,Reetz选择的突变残基大部分都在距离催化残基 以内的。与LipA相比较,CalB在分子量和结构复杂度上都有一定的增加。如图8B,CalB由317个氨基酸组成,结构上除了中间有标准水解酶类的α/β折叠外,周围还有4个α螺旋和2个β折叠包裹(Xie,Y.et al.Enhanced enzyme kinetic stability by increasing rigidity within the active site[J].Journal of Biological Chemistry,2014,289(11):7994-8006.);另外也有一段由5个氨基酸(residues 142~146)形成的α螺旋覆盖在活性口袋上,由于这段螺旋太短,并未对活力带来太大影响;也有文献报道这段螺旋并不被看作为盖子,故这段螺旋可以被看作是形成盖子的过渡态。LIP1则在结构上不同于CalB,它在分子量和结构复杂度上又有了一次高度超越。如图8C,LIP1由534个氨基酸组成,除了结构内部水解酶类标准的α/β折叠外,在酶蛋白的周围还有12个α螺旋和5个β折叠包裹;另外它的活性口袋上方有一段由29个氨基酸(residues 65~94)形成α螺旋作为盖子,这个盖子使它在结构上具有两种构型(open和closed)。综上分析LipA、CalB和LIP1的结构特征,会发现它们是酶蛋白质在分子量和结构上从小到大、从简单到复杂的典型代表,它们催化残基丝氨酸 以内高柔性氨基酸突变显著提高酶的稳定性,预示着“酶活性中心稳定化”策略存在一定通用性;从它们提高的结果中发现一些潜在的规律可能为其他酶类的稳定性改造带来重要指导信息。
实施例12、分析活性中心热点氨基酸的分布特征
对LipA、CalB和LIP1的稳定性提高结果与突变残基到催化残基的距离、相对B-factor值(以各自催化残基 内最高的B-factor值为参照,计算出其余单点突变残基的B-factor)进行了详细分析。用 表示所有单点突变提高稳定性的参数,其余负突变或组合突变都视为稳定性没有发生变化,在此基础上得到了图9的结果。发现距离催化残基 的范围内都有提高酶的稳定性,其中多数突变位于 且在 附近提高的幅度最为明显;而低于 和大于 的范围内未发现有单点氨基酸的突变提高酶稳定性;而相对B-factor在宽的分布范围内对酶稳定性的提高都有影响,主要集中在相对B-factor值60~70和90~103之间;相对B-factor值大于103和小于60未发现有单点突变提高酶稳定性,因此,“酶活性中心稳定化”策略在选取距离催化残基 和相对B-factor值60~100(以活性中心 内最高B-factor为参考)之间的高柔性残基进行突变,将会进一步提高酶稳定性改造的成功率。
一种高效提高酶热动力学稳定性的通用策略专利购买费用说明
![]()
Q:办理专利转让的流程及所需资料
A:专利权人变更需要办理著录项目变更手续,有代理机构的,变更手续应当由代理机构办理。
1:专利变更应当使用专利局统一制作的“著录项目变更申报书”提出。
2:按规定缴纳著录项目变更手续费。
3:同时提交相关证明文件原件。
4:专利权转移的,变更后的专利权人委托新专利代理机构的,应当提交变更后的全体专利申请人签字或者盖章的委托书。
Q:专利著录项目变更费用如何缴交
A:(1)直接到国家知识产权局受理大厅收费窗口缴纳,(2)通过代办处缴纳,(3)通过邮局或者银行汇款,更多缴纳方式
Q:专利转让变更,多久能出结果
A:著录项目变更请求书递交后,一般1-2个月左右就会收到通知,国家知识产权局会下达《转让手续合格通知书》。
动态评分
0.0